Origem da tradução das 4 bases de ácido nucleico para os 20 aminoácidos, e a atribuição de codons para Aminoácidos
http://elohim.heavenforum.org/t186-origem-da-traducao-das-4-bases-de-acido-nucleico-para-os-20-aminoacidos-e-a-atribuicao-de-codons-para-aminoacidos
A célula converte a informação transportada em uma molécula de mRNA ( RNA mensageiro ) numa molécula de proteína. Essa façanha de tradução foi um foco de atenção de biólogos no final dos anos 1950, quando ele se colocou como "problema de codificação": como funciona a tradução da informação em uma seqüência linear de nucleotídeos no RNA para a sequência linear de um conjunto quimicamente muito diferente de unidades de aminoácidos em proteínas?
O primeiro cientista depois de Watson e Crick a encontrar uma solução para o problema de codificação, que é a relação entre a estrutura do DNA e RNA, e a síntese de proteínas, foi físico russo George Gamov. Gamow publicou na edição de outubro 1953 da revista Nature uma solução chamada de "código de diamante", um código tripleto de sobreposição com base em um esquema de combinatória em que 4 nucleótidos arranjados 3-em-um devem especificar 20 aminoácidos. Um pouco como um idioma, este código altamente restritivo foi principalmente hipotético, baseado no conhecimento então atual do comportamento dos ácidos nucleicos e proteínas. 3
O conceito de codificação aplicada a especificidade genética foi um pouco enganador, como a tradução entre as quatro bases de ácido nucleico e os 20 aminoácidos obedeceria as regras de uma cifra em vez de um código. Como Crick reconheceu anos mais tarde, na análise linguística, cifras geralmente operam em unidades de comprimento regular (como no esquema de DNA tripleto), contrastando os códigos que operam em unidades de comprimento variável (por exemplo, palavras, frases). Mas a metáfora código funcionou bem, apesar de ter sido literalmente imprecisa, e nas palavras de Crick "," código genético "soa muito mais intrigante do que 'cifra genético'."
Uma sequência de mRNA é decodificado em conjuntos de três nucleótidos
Uma vez que o mRNA foi produzido por transcrição e processamento, as informações presentes na sua sequência nucleotídica é utilizada para sintetizar uma proteína. A transcrição é simples de compreender como um meio de transferência de informação: uma vez que DNA e RNA são química e estruturalmente semelhante, o DNA pode atuar como um molde para a síntese direta do RNA por emparelhamento de bases complementares. Como os significa transcrição prazo, é como se uma mensagem escrita à mão está sendo convertido, por exemplo, em um texto datilografado. A própria linguagem e forma de a mensagem não mudam, e os símbolos usados estão intimamente relacionados.
Em contraste, a conversão da informação no RNA em proteína representa uma tradução da informação numa outra língua que usa bastante diferentes símbolos. Além disso, uma vez que existem apenas 4 nucleótidos diferentes em mRNA e 20 tipos diferentes de aminoácidos de uma proteína, esta tradução não pode ser explicada por uma correspondência direta de um-para-um entre um nucleótido em RNA e um aminoácido na proteína. A sequência de nucleótidos de um gene, por intermédio de mRNA, traduz-se na sequência de aminoácidos de uma proteína. Este código foi decifrado no início de 1960.
Pergunta: como é que a tradução do triplet anti códon á aminoácidos, e sua atribuição, poderiam ter surgidos? Não existe afinidade entre o anti-códon físico e os aminoácidos. O que deve ser explicado, é o arranjo das "palavras" de códons na tabela códon padrão que é altamente não-aleatória, redundante e optimizada, e serve para traduzir os dados para a sequência de aminoácidos para fazer proteínas, e a origem do atribuição dos 64 codões tripleto para os 20 aminoácidos ? Isto é, a origem da sua tradução. A origem de um alfabeto através dos códigos tripletos é uma coisa, mas em cima, este código tem que ser traduzido para uma outro "alfabeto", constituído pelos 20 aminoácidos. Isso é como explicar a origem da capacidade de traduzir a linguagem Inglês para o chinês. Temos que constituir a linguagem e os símbolos Inglês e Chinês em primeiro lugar, a fim de conhecer a sua equivalência. O mesmo se refere a tradução da linguagem dos triplets, á linguagem dos amino ácidos. Só sabemos de processos mentais serem capazes de criar idiomas diferentes, e sua tradução.
Stephen Meyer, Signature in the cell, página 99:
nada acerca das características físicas ou químicas dos nucleótidos ou aminoácidos dita diretamente qualquer conjunto particular de atribuições entre aminoácidos e bases no texto do DNA. O código não pode ser deduzido a partir das propriedades químicas de amino ácidos e bases nucleotídicas. Assim como uma carta específica da língua Inglês pode ser representada por qualquer combinação de dígitos binários, de modo que também poderia um determinado aminoácido corresponder a qualquer combinação de bases de nucleótidos.
tRNA são moléculas adaptadoras. Um sistema de codificação ou tradução permite a tradução das informações a partir de sequências de bases de quatro caracteres de DNA para a "linguagem" de vinte caráteres ( amino ácidos ) de proteínas. a célula necessita de um meio para traduzir e expressar a informação armazenada no DNA. Grupos de três nucleótidos (chamados codões) no mRNA especificam a adição de um dos vinte aminoácidos formadores de proteína durante o processo de síntese de proteínas. Outros cientistas descobriram que a célula utiliza um conjunto de moléculas adaptadoras para ajudar a converter a informação do mRNA em proteínas.
Como é que surge especificidade e informação biológica funcional? As proteínas teriam que possuir as sequências corretas de aminoácidos, a fim de ser capazes de desbobinar e copiar informação genética; as proteínas e RNAs ribossomais teriam de ser sequenciadas com precisão, a fim de dobrar em subunidades que se encaixam em um conjunto para formar um ribossoma funcional; os RNAs de transferência teriam que intermediar as associações específicas, a fim de converter as sequências aleatórias de bases dos polinucleótidos em sequências de aminoácidos específicos; e as sequências de aminoácidos assim produzidos teriam que ser arranjados e organizados precisamente, a fim de dobrar em estruturas tridimensionais proteicas estáveis.
A sequência de nucleótidos da molécula de mRNA é lida em grupos consecutivos de três. O RNA é um polímero linear de quatro nucleótidos diferentes, de modo que existem 4 x 4 x 4 = 64 combinações possíveis de três nucleotídeos: os tripletos AAA, AUA, AUG, e assim por diante. No entanto, apenas 20 aminoácidos diferentes são comumente encontrados em proteínas. Ou alguns trigêmeos nucleotídeos nunca são usados, ou o código é redundante e alguns aminoácidos são especificados por mais de um tripleto. A segunda possibilidade é, de fato, a correta, como mostrado pelo código genético completamente decifrado mostrado abaixo:
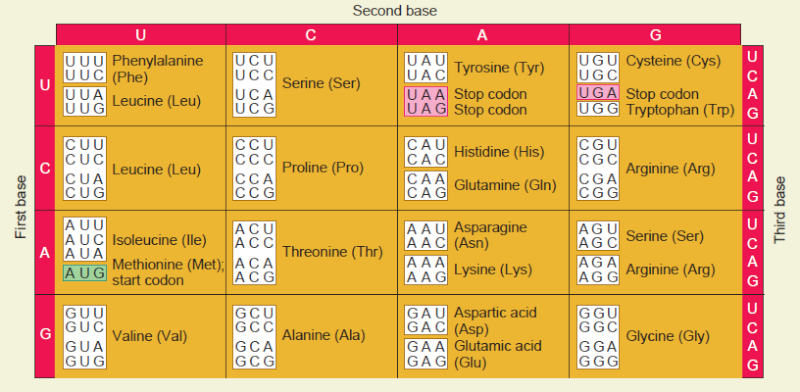
Cada grupo de três nucleótidos consecutivos em RNA é denominado um códon, e cada códon especifica um aminoácido ou uma paragem para o processo de tradução.
A princípio, uma sequência de RNA pode ser traduzida em qualquer um dos três enquadramentos de leitura diferentes, dependendo de onde começa o processo de descodificação (Figura abaixo).
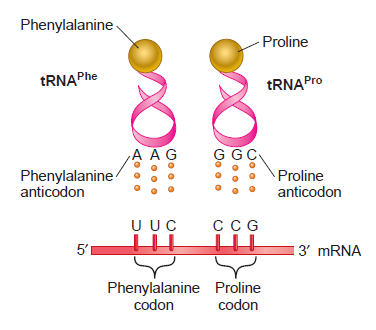
O códon de iniciação (AUG) define o quadro de leitura de um mRNA-uma sequência de códons determinada pela leitura bases em grupos de três, começando com o códon de iniciação. Este conceito é melhor entendido com alguns exemplos. A sequência de mRNA mostrado abaixo codifica um polipéptido curto com 7 aminoácidos:
No entanto, apenas uma das três possíveis grelhas de leitura de um mRNA codifica a proteína desejada. Para ilustrar isso, veja:

Se remover uma única base (C) adjacente ao códon de iniciação, isto altera a grelha de leitura para produzir um polipéptido com sequencia diferente:

Alternativamente, se nós removemos três bases (CCC) próximo ao códon de início , o polipeptídio resultante tem a mesma grelha de leitura do primeiro polipéptido, embora um aminoácido (Pro, prolina) tenha sido excluído:

Uma adição de um único nucleótido iria alterar o quadro de leitura para além do ponto de inserção, e abolir, assim, a função adequada da proteína codificada. Isto é chamado uma mutação frameshift, porque o mudou quadro de leitura. Além da mutação mediante adição ou remoção, pode haver uma mutação pontual, ou substituição, (o que tem mais efeitos adversos na sintetização de proteínas porque os nucleotídeos continuam a ser lidos em tripletos, mas em molduras diferentes - chamada de mutação "frameshift")
Em princípio, uma sequência de RNA pode ser traduzida em qualquer um dos três enquadramentos de leitura diferentes, dependendo de onde começa o processo de descodificação. No entanto, apenas um dos três possíveis grelhas de leitura de um mRNA codifica a proteína desejada. Um sinal de pontuação especial no início de cada mensagem de RNA que define a estrutura de leitura correta, no início da síntese de proteínas.
AUG é o códon universal de iniciação. Quase todos os organismos (e cada gene) que tem sido estudado usa a três ribonucleotídeos com a seqüência AUG para indicar o inicio da síntese de proteínas ( ponto de partida da tradução).
Pergunta: Por que e como processos naturais deveriam e teriam "escolhido" de inserir um sinal de pontuação, um códon de iniciação universal, para que o ribossomo pudesse "saber" aonde começar a tradução? Isto é essencial para que a máquina a começar a tradução no lugar correto. A única alternativa para uma programação inteligente seria o acaso formar esta sequencia. Mas e daí, como poderia em seguida o ribossomo ser programado a reconhecer o sinal de iniciação ? Perguntas sem respostas para quem quer propor mecanismos aleatórios naturais, sem inteligência envolvida. Isto é essencial para que a máquina a começar a tradução para o lugar correto.
Moléculas tRNA correspondem a aminoácidos de códons no mRNA
Os códons de uma molécula de mRNA não reconhecem diretamente os aminoácidos que especificam: o grupo de três nucleótidos , por exemplo, não se ligam diretamente aos aminoácidos. Em vez disso, a tradução do mRNA para proteínas depende de moléculas adaptadores que podem reconhecer e se ligar ambos ao códon e, em outro local na sua superfície, ao aminoácido. Estes adaptadores consistem de um conjunto de pequenas moléculas de RNA conhecidas como RNAs de transferência (tRNAs), cada uma com cerca de 80 nucleótidos de comprimento.
Características estruturais comuns são partilhadas entre todos os tRNAs
Para entender como tRNAs atuam como portadores dos amino ácidos corretos durante a tradução, os pesquisadores examinaram a estrutura das características destas moléculas em grande detalhe. Embora uma célula faz muitos tRNAs diferentes, todos compartilham características estruturais comuns. A estrutura secundária do tRNA exibe uma folha de trevo padrão. A tRNA tem três estruturas de haste-laçada, uma variável de poucos locais, e uma haste com um receptor. Regiões de cadeias simples . A haste aceitadora é um aminoácido, onde se torna anexado a um tRNA . Um sistema de numeração convencional para os nucleótidos dentro de uma molécula de tRNA começa na extremidade final 5' e prossegue para a extremidade 3' Entre os diferentes tipos de moléculas de tRNA, os sites de variáveis (em azul) podem diferir no número de nucleótidos que contêm. O anticódon está localizado na região do segundo gancho. A estrutura tridimensional, ou terciária do tRNA envolve a dobragem adicional de moléculas da estrutura secundária.
Na estrutura terciária do tRNA, as regiões de haste-laçada estão dobradas em uma molécula muito mais compacta. A capacidade de moléculas RNA de formar estruturas de haste-ansa e a dobragem terciária das moléculas tRNA são descritas no Capítulo 9 (ver Figura 1.19). Curiosamente, para além dos nucleotídeos normais A, U, G, e C,
moléculas de tRNA geralmente contêm nucleotídeos modificados dentro de suas estruturas primárias. Entre muitas espécies diferentes, os pesquisadores descobriram que mais de 80 modificações diferentes de nucleotídeos podem ocorrer em moléculas de tRNA.
Aminoacyl-tRNA Synthetases anexam os amino ácidos apropriados ao tRNAs
Para funcionar corretamente, cada tipo de tRNA deve ter o aminoácido apropriado ligado à sua extremidade 3 '. Como é que um aminoácido se liga a um tRNA com o anticódon correto? enzimas denominadas aminoacil-tRNA sintetases catalisam a fixação de aminoácidos á moléculas de tRNA. Células produzem 20 enzimas aminoacil-ARNt sintetase diferentes, 1 para cada um dos 20 aminoácidos distintos. Cada aminoacil-ARNt sintetase é nomeado para o aminoácido específico que atribui ao tRNA. Por exemplo, alanil-tRNA sintetase reconhece um tRNA com anticodon-tRNA de alanina -e atribui uma alanina a ele. Aminoacil-tRNA sintetases catalisam uma reação química envolvendo três moléculas diferentes: um aminoácido, uma molécula de tRNA, e ATP ( a energia ná célula ). No primeiro passo da reação, uma sintetase reconhece um amino ácido específico e também ATP
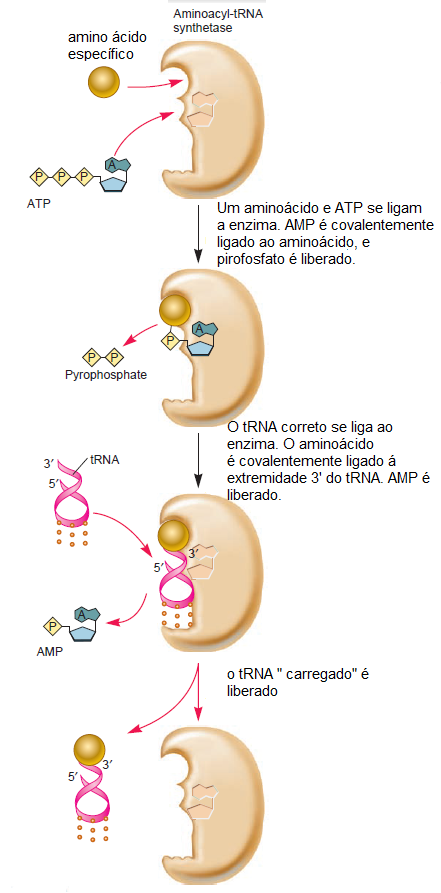
Figura 1.26.Função catalítica de aminoacyltRNA synthetase. Aminoacil-ARNt-sintetase tem locais de ligação para um amino ácido específico , ATP, e uma determinada tRNA. No primeiro passo, a enzima catalisa a ligação covalente de AMP a pelo amino ácido, obtendo-se até ao aminoácido ativado. No segunda passo, o aminoácido ativado é ligado ao tRNA apropriado.
o ATP é hidrolisado, e AMP se anexa ao amino ácido, e pirofosfato é liberado. Durante o segundo passo, o tRNA correto se liga a sintetase. O aminoácido se liga covalentemente à extremidade 3' da tRNA na haste aceitante, e AMP é liberado. Finalmente, o tRNA com seu aminoácido ligado é libertado da enzima. Nesse estágio,
o tRNA é chamado um tRNA carregado ou uma aminoacil-tRNA. Em uma molécula tRNA carregada, o aminoácido é ligado à extremidade 3' do tRNA por uma ligação covalente ( veja a inserção na Figura 1,25).
A capacidade das aminoacil-tRNA-sintetases de reconhecer tRNAs por vezes tem sido chamado de "segundo código genético." Este processo de reconhecimento é necessário para manter a fidelidade da informação genética. A freqüência de erro para aminoacil-tRNA sintetases é inferior a 10^5. Em outras palavras, o amino ácido errado está ligado a um tRNA é menos de uma vez em 100.000 vezes! Como poderia-se esperar, a região do anticódon do tRNA é normalmente importante para o reconhecimento preciso pela aminoacil-tRNA sintetase correta. Em estudos de sinthetases na bactéria Escherichia coli , 17 das 20 tipos de aminoacil-ARNt-sintetases reconhecem a região do anticódon do tRNA. No entanto, outras regiões do tRNA são igualmente reconhecidas. Os locais de reconhecimento são importantes. Estes incluem a haste aceitadora e
bases nas regiões haste de laço.
O tRNA e ATP encaixam precisamente no sítio ativo da enzima, e a estrutura é configurada e concebida para funcionar de uma forma finamente sintonizada. Como poderia um dispositivo tão funcional ser o resultado de forças aleatórias não guiados e reações químicas sem um objetivo final? Como o encaixe preciso para cada uma das vinte amino ácidos poderia ter surgido aleatoriamente, assim como o encaixe para a molécula ATP, que é indispensável para fornecer a energia necessária para a reação poder ocorrer ? Ademais, a aminoacil-tRNA-sintetases é muito específica para esta função, e não tem função nenhuma por si só, mas apenas, se devidamente encaixada no lugar certo para exercer a função especifica com exatidão.
Descasamentos que seguem a Regra Wobble podem ocorrer na terceira posição de emparelhamento no códon-anticódon
Depois de considerar a estrutura e função de moléculas de tRNA, vamos reexaminar algumas características sutis do código genético. O código genético é degenerado, o que significa que mais do que um códon pode especificar o mesmo aminoácido. Degeneração geralmente ocorre na terceira posição do códon. Por exemplo, valina é especificada por GUU, GUC, GUA, e GUG. Em todos os quatro casos, as primeiras duas bases são G e U. A terceira base, no entanto, pode ser U, C, A, ou G. Para explicar esse padrão de degeneração, Francis Crick propôs em 1966 que é devido a "oscilação" na terceira posição no processo de reconhecimento códon-anticódon. Segundo às regras de oscilação, nas duas posições, o primeiro par é estritamente de acordo à regra AU/GC . No entanto, a terceira posição pode tolerar certos tipos de descasamentos (Figura 1.27)
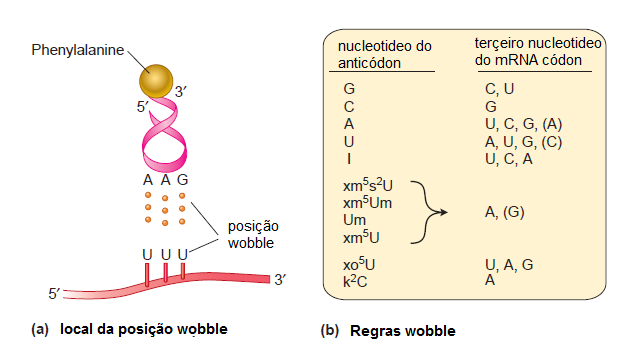
Figura 1.27 Posição Wobble e regras de emparelhamento de bases
(a) A posição oscilante ocorre entre a primeira base (ou seja, a primeira base na direção 5 'para 3') no
anticódon e a terceira base no códon do mRNA.
(b) As bases padrão encontradas no RNA são G, C, A, e U. Além disso, as estruturas das bases de tRNA podem ser modificadas. Algumas bases modificadas que podem ocorrer na posição de oscilação na tRNA são I = inosina; xm5s2U = 5-metil- 2-tiouridina; xm5Um = 5-metil-2'-O-metiluridina; Hum= 2'-O-metiluridina; xm5U = 5-metiluridina; xo5U =5-hydroxyuridine; K2C = lysidine (um derivado de citosina). As bases de mRNA em parênteses são muito mal reconhecidas pelo tRNA.
Por causa das regras de oscilação, é observada uma certa flexibilidade no reconhecimento entre um códon e anticódon durante o processo de tradução. Quando dois ou mais tRNA's que diferem na base oscilante são capazes de reconhecer o mesmo códon, estes são denominados isoacceptor tRNA. Como um exemplo, os tRNA's
com um anticódon de 3'-5'-CCA ou CCG-3'-5' podem reconhecer um codon com a sequência de 5'-3'-GGU. Além disso, a oscilação normas permitem que um único tipo de tRNA possa reconhecer mais que um códon. Por exemplo, um tRNA com uma sequência anticódon de 3'-5'-AAG pode reconhecer um 5'-UUC-3'-5'e um UUU-3'códon. O códon 5'-UUC-3'é uma combinação perfeita com este tRNA. O 5'-UUU-3'códon é incompatível de acordo com a norma. Regras de RNA-RNA de hibridação (ou seja, G no anticódon é incompatível para U no códon), mas os dois podem encaixar de acordo com a as regras de oscilação descritas na Figura 1.27. Da mesma forma, a modificação da base de oscilação para uma inosina permite reconhecer um tRNA de três codons diferentes. Ao nível celular, a capacidade de um tRNA único de reconhecer mais que um códon torna desnecessário de uma célula fazer 61 diferentes moléculas de tRNA com anticódons que são complementares às 61 possíveis códons sentidos. Células de E. coli, por exemplo, produzem uma população de moléculas de tRNA que tem apenas 40 diferentes sequências anticódon.
Moléculas de RNA podem dobrar em estruturas tridimensionais precisas, e as moléculas de tRNA fornecem um exemplo marcante. Quatro segmentos curtos das tRNA dobradas são de dupla hélice, a produção de uma molécula que se parece com uma folha de trevo quando desenhado esquematicamente. Veja abaixo:
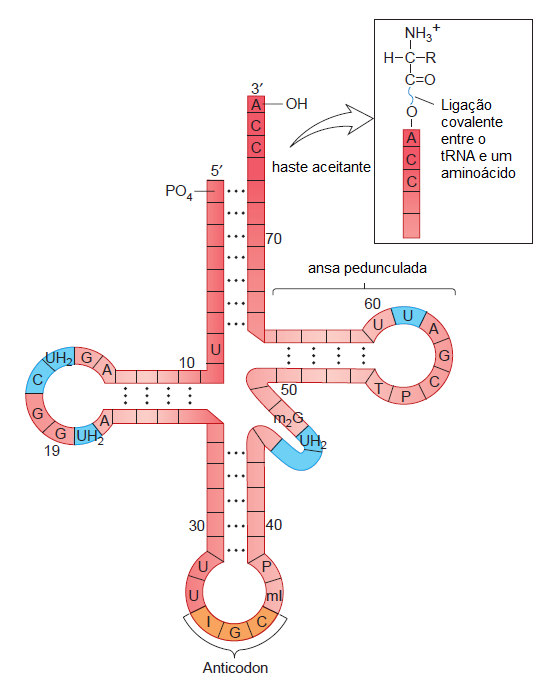
Figura 1.25 Estrutura Secundária de tRNA. A numeração convencional dos nucleotídeos inicia na extremidade 5 ' e procede para a extremidade 3 '. Em todos os tRNA's, os nucleotídeos na extremidade 3 'contêm a sequência CCA. Certos locais podem ter nucleótideos adicionais não encontrados em todas as moléculas de tRNA. Estes sítios variáveis são mostrados em azul. A figura também mostra as localizações de poucas bases modificadas especificamente encontradas num tRNA de levedura que transporta alanina. As bases modificadas são de seguinte forma: I = inosina, MI = metilinosina, T = ribothymidine, UH2 =dihydrouridine, M2G = dimethylguanosine, e P = pseudouridina. A inserção mostra um aminoácido ligado de forma covalente à extremidade 3 'de um tRNA.
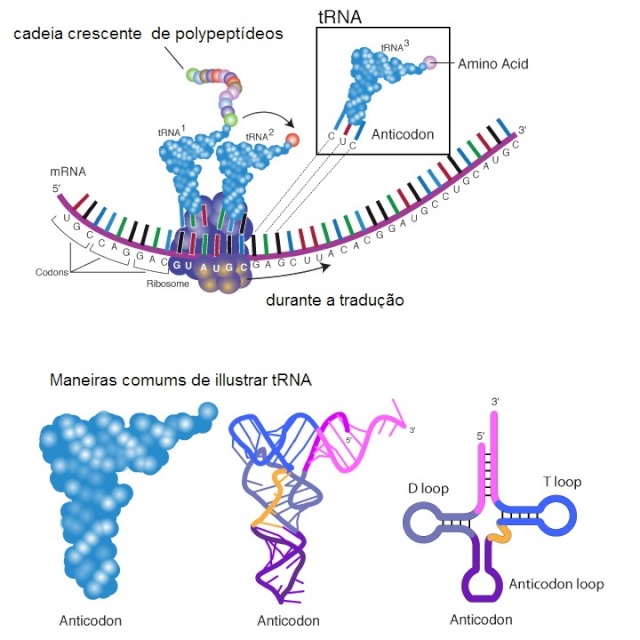
O reconhecimento do códon é ditado apenas pelo anticódon no tRNA; a estrutura química do aminoácido ligado ao tRNA não desempenha nenhum um papel.
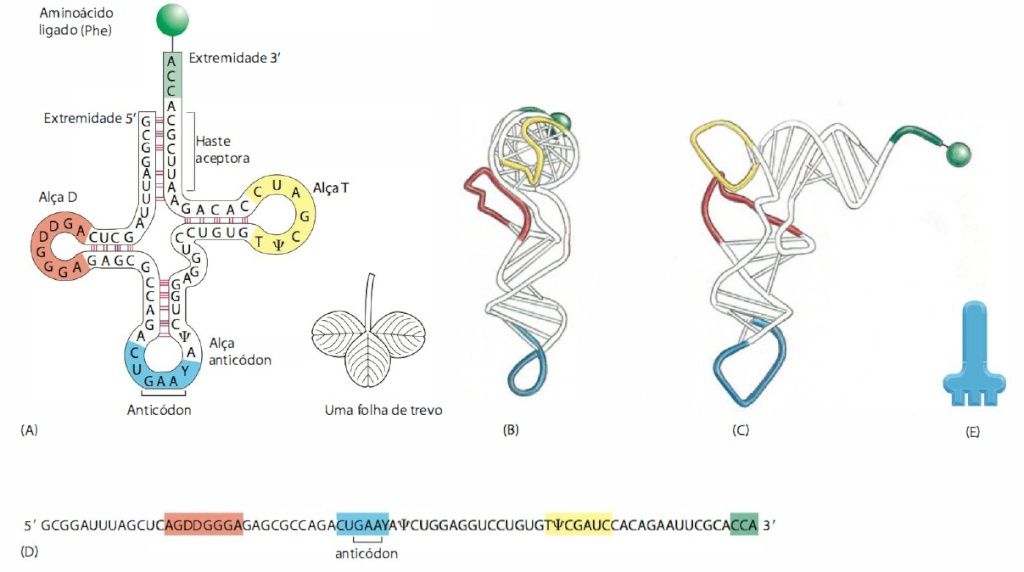
Uma molécula de tRNA. Um tRNA específico para o aminoácido fenilalanina (Phe) é ilustrado de várias maneiras. (A) A estrutura na forma de folha de trevo mostrando a complementaridade do pareamento de bases (linhas vermelhas) que cria as regiões de dupla-hélice na molécula. O anticódon é a sequência de três nucleotídeos que forma pares de bases com o (ódon no mRNA. O aminoácido correspondente ao par códon-anticódon é ligado na extremidade 3' do tRNA. Os tRNAs contêm algumas bases incomuns, as quais são produzidas por modificação química após a síntese do tRNA. Por exemplo, as bases identificadas como 'I' (pseudouridina) e D (diidrouridina) são derivadas de uracila. (B e C) Vistas da molécula em forma de L, com base em análise de difração de raios X. Embora este diagrama ilustre um tRNA para o aminoácido fenilalanina, todos os outros tRNAs têm estruturas semelhantes. (D) A sequência nucleotídica linear da molécula, colorida de acordo com (A), (B) e (C). (E) A representação do tRNA usada neste livro.
Por exemplo, uma sequência 5 "-GCUC-3" em uma parte de uma cadeia de um polinucleótido pode formar uma associação relativamente forte com um "-GAGC-3" sequência 5 na outra região da mesma molécula de tRNA. A folha de trevo é submetida a dobragem adicional, para formar uma estrutura em forma de L compacta que é mantida unida por ligações de hidrogênio adicionais entre as diferentes regiões da molécula. Duas regiões de nucleótidos desemparelhados situadas de um lado da molécula em forma de L são cruciais para a função do tRNA na síntese de proteínas. Uma destas regiões constitui o anticódon, um conjunto de três nucleótidos consecutivos que forma o par ( chave e fechadura ) com o códon complementar de uma molécula de mRNA. A outra é uma região única curta de cadeia simples no "fim da molécula , isto é o local onde o aminoácido que corresponde ao códon se liga ao tRNA. O código genético é redundante;. Isto é, vários códons diferentes podem especificar um único aminoácido. Esta redundância implica que existam mais de um tRNA para muitos dos aminoácidos ou que algumas moléculas de tRNA podem formar um par ( chave na fechadura ) com bases, com mais do que um códon. De fato, ambas as situações ocorrem. Alguns aminoácidos tem mais de um tRNA e alguns tRNA's são construídos de modo que eles necessitam de um emparelhamento de bases preciso apenas com as duas primeiras posições do códon e podem tolerar uma incompatibilidade (ou oscilar) na terceira posição.
A edição por tRNA-sintetases assegura a exatidão
Vários mecanismos trabalhando em conjunto asseguram que a tRNA-sintetase ligue o aminoácido correto a cada tRNA. A sintetase deve inicialmente selecionar o aminoácido correto e o faz, principalmente, por meio de um mecanismo composto de duas etapas. Primeiro, o aminoácido correto apresenta uma maior afinidade pela fenda do sítio ativo de sua sintetase, sendo assim favorecido sobre os outros 19. Em particular, os aminoácidos maiores do que o correto são efetivamente excluídos do sítio ativo. No entanto, a discriminação exata entre dois aminoácidos semelhantes, como a isoleucina e a valina (os quais diferem apenas por um grupo metil), é muito difícil de ser alcançada por um mecanismo de reconhecimento de etapa única. Uma segunda etapa de discriminação ocorre após o aminoácido ter sido ligado covalentemente ao AMP (Figura 1.28)
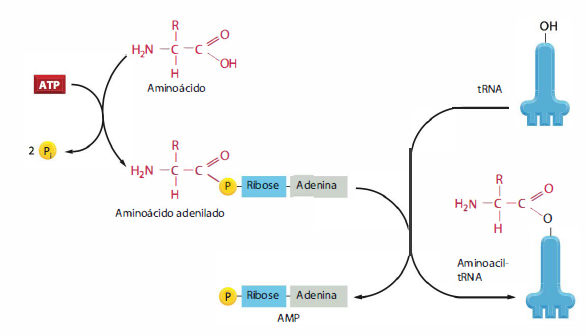
Figura 1.28 Ativação do aminoácido.
Um aminoácido é ativado para a síntese proteica por uma enzima aminoacil-tRNA-sintetase em duas etapas. Como indicado, a energia da hidrólise de ATP é usada para ligar cada aminoácido a sua molécula de tRNA em uma ligação altamente energética. ° aminoácido é inicialmente ativado por meio da ligação de seu grupo carboxila diretamente a um AMP, formando um aminoácido adenilado; a ligação do AMP, normalmente uma reação desfavorável, é dirigida pela hidrólise da molécula de ATP que doa o AMP. Sem deixar a enzima sintetase, o grupo carboxila ligado ao AMP no aminoácido é, então, transferido para um grupo hidroxila no açúcar, na extremidade 3' da molécula de tRNA. Essa transferência liga o aminoácido por meio de uma ligação éster ativada ao tRNA, formando a molécula
final de aminoacil-tRNA. A enzima sintetase não é mostrada neste diagrama.
Quando o tRNA liga-se à sintetase, ele empurra o aminoácido para uma segunda fenda na sintetase, cujas dimensões precisas excluem o aminoácido correto, mas permitem o acesso de aminoácidos intimamente relacionados. Uma vez que um aminoácido entre nessa fenda de edição, ele será hidrolisado do AMP (ou do próprio tRNA, caso a ligação aminoacil-tRNA já tenha sido formada) e será liberado da enzima. Essa edição hidrolítica, a qual é análoga à correção exonucleotídica mediada pelas DNA-polimerases (Figura 1.29) eleva a exatidão média da taxa de carregamento de tRNA para aproximadamente um erro a cada 40 mil acoplamentos.
Alta fidelidade
Aminoacil-ARNt sintetases devem executar as suas tarefas com alta precisão. Cada erro resultará num aminoácido deslocado quando novas proteínas são construídos. Essas enzimas fazem apenas um erro em 10.000. Para a maioria dos aminoácidos, este nível de precisão não é muito difícil de alcançar. A maioria dos aminoácidos são muito diferentes uns dos outros, e, muitas partes das diferentes tRNA's são utilizadas para reconhecimento preciso. Mas em alguns casos, é difícil escolher apenas os aminoácidos corretos e estas enzimas devem recorrer a técnicas especiais.
Isoleucina é particularmente difícil de reconhecer. É reconhecida por um furo em forma de isoleucina na enzima, o qual é muito pequeno para caber aminoácidos maiores como metionina e fenilalanina, é muito hidrófobo, para ligar qualquer coisa com cadeias laterais polares. Mas, a pouco menor aminoácido valina, diferente por apenas um único grupo metilo, também se encaixa muito bem neste bolso, a ligação em vez de isoleucina ocorre cerca 1 em cada 150 vezes. É demasiado muitos erro, por isso devem ser tomadas medidas corretivas. Isoleucil-tRNA sintetase resolve este problema com um segundo sítio ativo, que realiza uma reação de edição. Isoleucina não se encaixa neste sitio, mas valina errante o faz. O erro é então clivadao, deixando o tRNA pronto para um aminoácido leucina devidamente colocado. Esta etapa de correção melhora a taxa de erro global de cerca de 1 em 3.000.
Esta é uma técnica incrível de revisão de erro, que se junta a outros incríveis mecanismos super elaborados de reparo na célula. Mais uma vez surge a pergunta: Como é que estas máquinas moleculares precisas poderiam ter surgido por meios naturais, sem inteligência envolvido? Isto parece-me mais um incrível exemplo de nano maquinaria molecular altamente sofisticada projetada para desempenhar a sua missão com alto grau de fidelidade e minimização de erros, o que só pode surgir pela previdência de um criador incrivelmente inteligente.
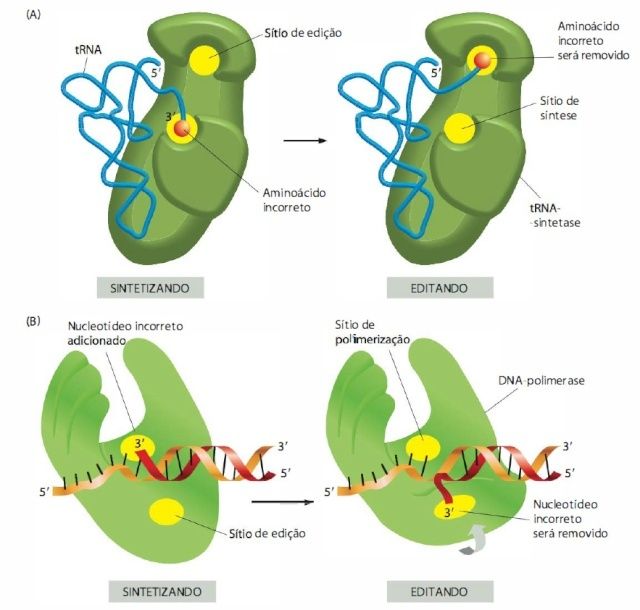
Figura 1.29 Edição hidrolítica. (A) A tRNA-sintetase remove seus próprios erros de pareamento por meio da edição hidrolítica de aminoácidos incorporados incorretamente. O aminoácido correto é rejeitado pelo sitio de edição. (B) ° processo de erro correção realizado pela DNA-polimerase apresenta algumas similaridades; entretanto, difere no que diz respeito ao processo de remoção, o qual depende fortemente de um erro de pareamento com o molde.
Papel crucial das enzimas Aminoacil-tRNA sintetases que ativam o tRNA
Para tentar explicar a origem do código genético, vários pesquisadores têm procurado algum tipo de afinidade química entre os aminoácidos e seus códons correspondentes. Mas esta abordagem é equivocada:
Em primeiro lugar, o código é mediado por tRNA que tem o anticódon (do mRNA) em vez do próprio códon (no DNA). Portanto, se o código fosse baseado em afinidades entre os aminoácidos e os anticódons, isso implicaria que o processo de tradução, através de transcrição não poderia ter surgido em uma segunda fase ou melhoria em um sistema direto mais simples - o complexo processo de duas etapas seria necessário ter surgido desde o início.
Em segundo lugar, o aminoácido não tem um papel na identificação do tRNA ou o códon. Esta associação é feita pela enzima de ativação Aminoacil-ARNt sintetase, que atribui a cada um dos aminoácidos o tRNA apropriado (que requer claramente a enzima identificar corretamente os dois componentes). Existem diferentes enzimas de ativação, 20 - um para cada tipo de aminoácido.
Curiosamente, o lugar do tRNA à qual o aminoácido é anexado tem a mesma sequência nucleotídica para todos os aminoácidos - que constitui um terceiro motivo.
O interesse pelo código genético tende a concentrar-se sobre o papel do tRNA, mas como apenas mencionado, isso é apenas a metade da tarefa de implementação do código para formar proteínas. Tão importante como o emparelhamento códon -anticódon (entre mRNA e tRNA) é a capacidade de cada uma das enzimas de ativação de reunir e anexar cada aminoácido ao seu tRNA apropriado. É evidente que a aplicação do código requer dois conjuntos de moléculas intermediárias: os tRNAs que interagem com os ribossomos e reconhecem o códon apropriado no mRNA, e as enzimas de ativação, Aminoacil-ARNt sintetases, que ligam e anexam os aminoácidos diretamente ao tRNA correto mediante o encaixe preciso e correto ( como se fosse chave-fechadura) . Este é o tipo de complexidade que permeia os sistemas biológicos, e que representa um desafio formidável para uma explicação naturalista de passo a passo mediante eventos apenas baseados em afinidade quimica, ou sorte, para a sua origem. Seria improvável o suficiente se o código foi implementado apenas pelos tRNAs que têm de 70 a 80 nucleotídeos; mas o papel igualmente crucial e complementar os amino ácidos corretos com das enzimas de ativação, que tem centenas de aminoácidos de comprimento. Isto exclui qualquer possibilidade realista de que esse tipo de arranjo poderia ter surgido de forma oportunista, de " boa sorte ", ou acaso. Evolução não seria também um mecanismo viável neste estagio, pois a evolução depende desse processo para começar a ocorrer. Estes mecanismos tinham que surgir antes da primeira célula começar a se auto-replicar.
O desenvolvimento progressivo do código genético não é realista
Tendo em vista os muitos componentes envolvidos na implementação do código genético, pesquisadores da origem da vida tentaram ver como ele poderia ter surgido de forma gradual, de maneira evolutiva. Por exemplo, é geralmente sugerido que o código genético começou com a aplicação de apenas alguns aminoácidos, que, em seguida, gradualmente aumentaram em número. Mas esse tipo de cenário encontra todos os tipos de dificuldades com algo tão fundamental como o código genético.
Em primeiro lugar, afigura-se que os primeiros códons precisavam ter usado apenas duas bases (que pode codificar até 16 aminoácidos); mas uma alteração posterior a três bases (para acomodar 20) iria perturbar gravemente o código. Reconhecendo esta dificuldade, a maioria dos investigadores assumiram que o código utilizava códons de 3 bases, desde o início; que foi notavelmente fortuito ou implicaria em alguma medida de previsão por parte da evolução (o que, evidentemente, não é permitido).
Muito mais grave são as implicações para as proteínas com base em um conjunto severamente limitado de aminoácidos. Em particular, se o código foi limitado a apenas alguns aminoácidos, em seguida, deve-se presumir que as enzimas de ativação composto apenas esse conjunto limitado de aminoácidos, e ainda tinham o nível necessário de especificidade para a implementação segura do código. Não há nenhuma evidência disso; e subsequente a reorganização das enzimas que fariam uso de aminoácidos recentemente disponíveis exigiria mudanças altamente improváveis em sua configuração. Limitações semelhantes se aplicaria aos componentes proteicos dos ribossomos, que têm um papel igualmente essencial na tradução.
Além disso, os tRNA's tendem a ter bases atípicas que são sintetizadas da maneira usual, mas posteriormente modificadas. Estas modificações são realizadas por enzimas, de modo que estas enzimas também teriam de ter começado a vida com base num número limitado de aminoácidos; ou tem de se supor que essas modificações são refinamentos posteriores - mesmo que eles parecem ser necessários para a execução segura do código.
Finalmente, o que vai motivar a adição de novos aminoácidos para o código genético? Eles teriam pouca ou nenhuma utilidade até incorporados em proteínas - mas isso não iria acontecer até que fossem incluídas no código genético. Assim, os novos aminoácidos deve ser sintetizados e de alguma forma incorporados em proteínas úteis (por enzimas que eles não têm), e toda a maquinaria necessária para incluí-los no código (tRNAs dedicados e ativando enzimas) postas em prática - e tudo feito de forma oportunista! Totalmente incrível!
O que deve ser explicado, é o arranjo dos códons na tabela códons padrão que é altamente não aleatório, e serve para traduzir a informação genetica na sequência de aminoácidos para fazer proteínas, bem como a origem da atribuição dos 64 codões tripleto para o 20 aminoácidos. Isto é, a origem da sua tradução. A origem de um alfabeto através dos códons triplet é uma coisa, mas em cima, tem que ser traduzido para um outro "alfabeto", constituído pela sequência de 20 aminoácidos. Isto é, como explicar a origem da capacidade de traduzir a linguagem Inglês para o chinês. Em cima disso, a origem da própria máquina para promover o processo em si tem também que ser explicada, que é o hardware. Quando os seres humanos traduzem Inglês para o chinês, por exemplo, nós reconhecemos a palavra Inglês, e o tradutor conhece o símbolo chinês equivalente e o escreve. Na célula, enzimas Aminoacil-ARNt-sintetase reconhecem o anticódon do tRNA tripleto, e anexam o aminoácido equivalente correto ao tRNA, formando assim cadeias proteicas, que no final faz surgir um polipeptídeo, que se dobra em uma proteína funcional. Como poderiam reações químicas aleatórias ter produzido esse reconhecimento?
Se um codon/anticodon/tRNA não for atribuído ao Aminoacil-ARNt-sintetase correto, o que aconteceria? Vamos supor que o códon AAA em vez de ser atribuído ao amino ácido lisina, for atribuido ao amino ácido aspargina, a proteína vai dobrar de forma errada, ou não vai dobrar, e não vai ser funcional, e vai para a proteassoma, a lixeira celular. O que você acha, como surgiu a atribuição correta do código genético de códons aos amino ácidos corretos ? Se você tiver o alfabeto, e precisar fazer a tradução para o chines, será que com tentativa/erro dos caracteres em chinês, você um dias vai conseguir formular uma frase compreensível em chinês ? as possibilidades são inúmeras, praticamente infinitas. E enquanto a atribuição correta não estava surgindo, a célula ficava esperando num cantinho tomando um Whisky ? Algumas teorias tentam explicar o mecanismo, mas todas elas continuam a ser insatisfatórias. Obviamente. Além disso, Aminoacil-ARNt-sintetase são enzimas complexas.
Por que razão é que elas teriam surgidas, se a função final só poderia ser exercida depois de todo o processo de tradução tiver sido implantado no local, com um ribossomo totalmente funcional ser capaz de fazer o seu trabalho? Lembrando a situação catch22, uma vez que pelo próprio ribossomo é feito o processo próprio em questão? Por que não é racional concluir que o próprio código, o software, bem como o hardware, sua origem são melhor explicados por meio da invenção de um ser extraordinariamente inteligente, em vez de afinidades e reações químicas aleatórias? Perguntas: que utilidade teria o ribossomo sem o tRNA ? sem aminoácidos ? - que são o produto de processos químicos evias metabólicas extremamente complexos ? Que utilidade teria a maquinaria toda, se o código não estivesse estabelecido ( também codificando a construção das maquinas como os ribossomos em questão ? ) , e se nem a atribuição de cada códon ao respectivo aminoácido estivesse presente ? tinha o software e o hardware não ter que estar no lugar ao mesmo tempo?
Haviam todas as partes não ter que estar totalmente funcional plenamente desenvolvidas, entreligados, completamente montados, e ajustados para exercer seu trabalho com precisão como um relógio? E mesmo que vamos supor, as peças estariam totalmente funcionais e no lugar, que vantagem teria isso, se todas as outras peças necessárias não estariam no lugar também, isto é, a dupla hélice do DNA, a sua compactação através de histonas e cromatinas e cromossomos, o seu mecanismo altamente complexo de extração de informação e a transcrição em mRNA? Tinha todo o processo, que é a iniciação da transcrição,o alongamento, o splicing, poliadenilação e terminação, exportação do núcleo para o citosol, a iniciação da síntese de proteínas (TRADUÇÃO), a conclusão da síntese de proteína e DOBRADURA correta das proteínas mediante chaperones , e sua respectiva máquinaria não ter que estar toda no lugar? Será que isso não constitui um complexo sistema interdependente, e irredutível?
Um aspecto raramente discutido de complexidade é como os tRNA's são designados para os aminoácidos corretos. Para o idioma de DNA possa ser traduzido adequadamente, cada anticódon tRNA deve ser ligado ao aminoácido correto. Se este passo crucial na replicação do DNA não for funcional, então a linguagem do DNA não serve para nada. As enzimas aminoacil tRNA sintetases (aaRSs) garantem que o aminoácido apropriado é ligado a um tRNA com o anticódon correto através de uma reação química chamada ' aminoacylação". Tradução precisa exige não só que cada tRNA seja atribuído ao aminoácido correto, mas também que ele não seja aminoacilado por qualquer uma das moléculas de aminoacil tRNA sintetases (aaRSs) dos outros 19 aminoácidos. Um livro de bioquímica observa que, porque todos os aaRSs catalisam reações semelhantes sobre várias moléculas de tRNA semelhantes, pensava-se que "evoluiram de um ancestral comum e devem, portanto, ser estruturalmente relacionados." (Voet e Voet pg. 971-975) No entanto, este não e o caso como "aaRSs formamd um grupo diversificado de [mais de 100] enzimas ... e há pouca semelhança de sequência entre synthetases específicas para os diferentes aminoácidos." (Voet e Voet pg 971-975). Surpreendentemente, estes próprios aaRSs são codificados pelo DNA: isto constitui a essência de um problema de ovo de galinha. As enzimas a construir ajudam a realizar a mesma tarefa que as constrói!
tradução de:
http://reasonandscience.heavenforum.org/t2057-origin-of-translation-of-the-4-nucleic-acid-bases-and-the-20-amino-acids-and-the-universal-assignment-of-codons-to-amino-acids#3528
http://elohim.heavenforum.org/t186-origem-da-traducao-das-4-bases-de-acido-nucleico-para-os-20-aminoacidos-e-a-atribuicao-de-codons-para-aminoacidos
A célula converte a informação transportada em uma molécula de mRNA ( RNA mensageiro ) numa molécula de proteína. Essa façanha de tradução foi um foco de atenção de biólogos no final dos anos 1950, quando ele se colocou como "problema de codificação": como funciona a tradução da informação em uma seqüência linear de nucleotídeos no RNA para a sequência linear de um conjunto quimicamente muito diferente de unidades de aminoácidos em proteínas?
O primeiro cientista depois de Watson e Crick a encontrar uma solução para o problema de codificação, que é a relação entre a estrutura do DNA e RNA, e a síntese de proteínas, foi físico russo George Gamov. Gamow publicou na edição de outubro 1953 da revista Nature uma solução chamada de "código de diamante", um código tripleto de sobreposição com base em um esquema de combinatória em que 4 nucleótidos arranjados 3-em-um devem especificar 20 aminoácidos. Um pouco como um idioma, este código altamente restritivo foi principalmente hipotético, baseado no conhecimento então atual do comportamento dos ácidos nucleicos e proteínas. 3
O conceito de codificação aplicada a especificidade genética foi um pouco enganador, como a tradução entre as quatro bases de ácido nucleico e os 20 aminoácidos obedeceria as regras de uma cifra em vez de um código. Como Crick reconheceu anos mais tarde, na análise linguística, cifras geralmente operam em unidades de comprimento regular (como no esquema de DNA tripleto), contrastando os códigos que operam em unidades de comprimento variável (por exemplo, palavras, frases). Mas a metáfora código funcionou bem, apesar de ter sido literalmente imprecisa, e nas palavras de Crick "," código genético "soa muito mais intrigante do que 'cifra genético'."
Uma sequência de mRNA é decodificado em conjuntos de três nucleótidos
Uma vez que o mRNA foi produzido por transcrição e processamento, as informações presentes na sua sequência nucleotídica é utilizada para sintetizar uma proteína. A transcrição é simples de compreender como um meio de transferência de informação: uma vez que DNA e RNA são química e estruturalmente semelhante, o DNA pode atuar como um molde para a síntese direta do RNA por emparelhamento de bases complementares. Como os significa transcrição prazo, é como se uma mensagem escrita à mão está sendo convertido, por exemplo, em um texto datilografado. A própria linguagem e forma de a mensagem não mudam, e os símbolos usados estão intimamente relacionados.
Em contraste, a conversão da informação no RNA em proteína representa uma tradução da informação numa outra língua que usa bastante diferentes símbolos. Além disso, uma vez que existem apenas 4 nucleótidos diferentes em mRNA e 20 tipos diferentes de aminoácidos de uma proteína, esta tradução não pode ser explicada por uma correspondência direta de um-para-um entre um nucleótido em RNA e um aminoácido na proteína. A sequência de nucleótidos de um gene, por intermédio de mRNA, traduz-se na sequência de aminoácidos de uma proteína. Este código foi decifrado no início de 1960.
Pergunta: como é que a tradução do triplet anti códon á aminoácidos, e sua atribuição, poderiam ter surgidos? Não existe afinidade entre o anti-códon físico e os aminoácidos. O que deve ser explicado, é o arranjo das "palavras" de códons na tabela códon padrão que é altamente não-aleatória, redundante e optimizada, e serve para traduzir os dados para a sequência de aminoácidos para fazer proteínas, e a origem do atribuição dos 64 codões tripleto para os 20 aminoácidos ? Isto é, a origem da sua tradução. A origem de um alfabeto através dos códigos tripletos é uma coisa, mas em cima, este código tem que ser traduzido para uma outro "alfabeto", constituído pelos 20 aminoácidos. Isso é como explicar a origem da capacidade de traduzir a linguagem Inglês para o chinês. Temos que constituir a linguagem e os símbolos Inglês e Chinês em primeiro lugar, a fim de conhecer a sua equivalência. O mesmo se refere a tradução da linguagem dos triplets, á linguagem dos amino ácidos. Só sabemos de processos mentais serem capazes de criar idiomas diferentes, e sua tradução.
Stephen Meyer, Signature in the cell, página 99:
nada acerca das características físicas ou químicas dos nucleótidos ou aminoácidos dita diretamente qualquer conjunto particular de atribuições entre aminoácidos e bases no texto do DNA. O código não pode ser deduzido a partir das propriedades químicas de amino ácidos e bases nucleotídicas. Assim como uma carta específica da língua Inglês pode ser representada por qualquer combinação de dígitos binários, de modo que também poderia um determinado aminoácido corresponder a qualquer combinação de bases de nucleótidos.
tRNA são moléculas adaptadoras. Um sistema de codificação ou tradução permite a tradução das informações a partir de sequências de bases de quatro caracteres de DNA para a "linguagem" de vinte caráteres ( amino ácidos ) de proteínas. a célula necessita de um meio para traduzir e expressar a informação armazenada no DNA. Grupos de três nucleótidos (chamados codões) no mRNA especificam a adição de um dos vinte aminoácidos formadores de proteína durante o processo de síntese de proteínas. Outros cientistas descobriram que a célula utiliza um conjunto de moléculas adaptadoras para ajudar a converter a informação do mRNA em proteínas.
Como é que surge especificidade e informação biológica funcional? As proteínas teriam que possuir as sequências corretas de aminoácidos, a fim de ser capazes de desbobinar e copiar informação genética; as proteínas e RNAs ribossomais teriam de ser sequenciadas com precisão, a fim de dobrar em subunidades que se encaixam em um conjunto para formar um ribossoma funcional; os RNAs de transferência teriam que intermediar as associações específicas, a fim de converter as sequências aleatórias de bases dos polinucleótidos em sequências de aminoácidos específicos; e as sequências de aminoácidos assim produzidos teriam que ser arranjados e organizados precisamente, a fim de dobrar em estruturas tridimensionais proteicas estáveis.
A sequência de nucleótidos da molécula de mRNA é lida em grupos consecutivos de três. O RNA é um polímero linear de quatro nucleótidos diferentes, de modo que existem 4 x 4 x 4 = 64 combinações possíveis de três nucleotídeos: os tripletos AAA, AUA, AUG, e assim por diante. No entanto, apenas 20 aminoácidos diferentes são comumente encontrados em proteínas. Ou alguns trigêmeos nucleotídeos nunca são usados, ou o código é redundante e alguns aminoácidos são especificados por mais de um tripleto. A segunda possibilidade é, de fato, a correta, como mostrado pelo código genético completamente decifrado mostrado abaixo:

Cada grupo de três nucleótidos consecutivos em RNA é denominado um códon, e cada códon especifica um aminoácido ou uma paragem para o processo de tradução.
A princípio, uma sequência de RNA pode ser traduzida em qualquer um dos três enquadramentos de leitura diferentes, dependendo de onde começa o processo de descodificação (Figura abaixo).

O códon de iniciação (AUG) define o quadro de leitura de um mRNA-uma sequência de códons determinada pela leitura bases em grupos de três, começando com o códon de iniciação. Este conceito é melhor entendido com alguns exemplos. A sequência de mRNA mostrado abaixo codifica um polipéptido curto com 7 aminoácidos:
No entanto, apenas uma das três possíveis grelhas de leitura de um mRNA codifica a proteína desejada. Para ilustrar isso, veja:

Se remover uma única base (C) adjacente ao códon de iniciação, isto altera a grelha de leitura para produzir um polipéptido com sequencia diferente:

Alternativamente, se nós removemos três bases (CCC) próximo ao códon de início , o polipeptídio resultante tem a mesma grelha de leitura do primeiro polipéptido, embora um aminoácido (Pro, prolina) tenha sido excluído:

Uma adição de um único nucleótido iria alterar o quadro de leitura para além do ponto de inserção, e abolir, assim, a função adequada da proteína codificada. Isto é chamado uma mutação frameshift, porque o mudou quadro de leitura. Além da mutação mediante adição ou remoção, pode haver uma mutação pontual, ou substituição, (o que tem mais efeitos adversos na sintetização de proteínas porque os nucleotídeos continuam a ser lidos em tripletos, mas em molduras diferentes - chamada de mutação "frameshift")
Em princípio, uma sequência de RNA pode ser traduzida em qualquer um dos três enquadramentos de leitura diferentes, dependendo de onde começa o processo de descodificação. No entanto, apenas um dos três possíveis grelhas de leitura de um mRNA codifica a proteína desejada. Um sinal de pontuação especial no início de cada mensagem de RNA que define a estrutura de leitura correta, no início da síntese de proteínas.
AUG é o códon universal de iniciação. Quase todos os organismos (e cada gene) que tem sido estudado usa a três ribonucleotídeos com a seqüência AUG para indicar o inicio da síntese de proteínas ( ponto de partida da tradução).
Pergunta: Por que e como processos naturais deveriam e teriam "escolhido" de inserir um sinal de pontuação, um códon de iniciação universal, para que o ribossomo pudesse "saber" aonde começar a tradução? Isto é essencial para que a máquina a começar a tradução no lugar correto. A única alternativa para uma programação inteligente seria o acaso formar esta sequencia. Mas e daí, como poderia em seguida o ribossomo ser programado a reconhecer o sinal de iniciação ? Perguntas sem respostas para quem quer propor mecanismos aleatórios naturais, sem inteligência envolvida. Isto é essencial para que a máquina a começar a tradução para o lugar correto.
Moléculas tRNA correspondem a aminoácidos de códons no mRNA
Os códons de uma molécula de mRNA não reconhecem diretamente os aminoácidos que especificam: o grupo de três nucleótidos , por exemplo, não se ligam diretamente aos aminoácidos. Em vez disso, a tradução do mRNA para proteínas depende de moléculas adaptadores que podem reconhecer e se ligar ambos ao códon e, em outro local na sua superfície, ao aminoácido. Estes adaptadores consistem de um conjunto de pequenas moléculas de RNA conhecidas como RNAs de transferência (tRNAs), cada uma com cerca de 80 nucleótidos de comprimento.
Características estruturais comuns são partilhadas entre todos os tRNAs
Para entender como tRNAs atuam como portadores dos amino ácidos corretos durante a tradução, os pesquisadores examinaram a estrutura das características destas moléculas em grande detalhe. Embora uma célula faz muitos tRNAs diferentes, todos compartilham características estruturais comuns. A estrutura secundária do tRNA exibe uma folha de trevo padrão. A tRNA tem três estruturas de haste-laçada, uma variável de poucos locais, e uma haste com um receptor. Regiões de cadeias simples . A haste aceitadora é um aminoácido, onde se torna anexado a um tRNA . Um sistema de numeração convencional para os nucleótidos dentro de uma molécula de tRNA começa na extremidade final 5' e prossegue para a extremidade 3' Entre os diferentes tipos de moléculas de tRNA, os sites de variáveis (em azul) podem diferir no número de nucleótidos que contêm. O anticódon está localizado na região do segundo gancho. A estrutura tridimensional, ou terciária do tRNA envolve a dobragem adicional de moléculas da estrutura secundária.
Na estrutura terciária do tRNA, as regiões de haste-laçada estão dobradas em uma molécula muito mais compacta. A capacidade de moléculas RNA de formar estruturas de haste-ansa e a dobragem terciária das moléculas tRNA são descritas no Capítulo 9 (ver Figura 1.19). Curiosamente, para além dos nucleotídeos normais A, U, G, e C,
moléculas de tRNA geralmente contêm nucleotídeos modificados dentro de suas estruturas primárias. Entre muitas espécies diferentes, os pesquisadores descobriram que mais de 80 modificações diferentes de nucleotídeos podem ocorrer em moléculas de tRNA.
Aminoacyl-tRNA Synthetases anexam os amino ácidos apropriados ao tRNAs
Para funcionar corretamente, cada tipo de tRNA deve ter o aminoácido apropriado ligado à sua extremidade 3 '. Como é que um aminoácido se liga a um tRNA com o anticódon correto? enzimas denominadas aminoacil-tRNA sintetases catalisam a fixação de aminoácidos á moléculas de tRNA. Células produzem 20 enzimas aminoacil-ARNt sintetase diferentes, 1 para cada um dos 20 aminoácidos distintos. Cada aminoacil-ARNt sintetase é nomeado para o aminoácido específico que atribui ao tRNA. Por exemplo, alanil-tRNA sintetase reconhece um tRNA com anticodon-tRNA de alanina -e atribui uma alanina a ele. Aminoacil-tRNA sintetases catalisam uma reação química envolvendo três moléculas diferentes: um aminoácido, uma molécula de tRNA, e ATP ( a energia ná célula ). No primeiro passo da reação, uma sintetase reconhece um amino ácido específico e também ATP

Figura 1.26.Função catalítica de aminoacyltRNA synthetase. Aminoacil-ARNt-sintetase tem locais de ligação para um amino ácido específico , ATP, e uma determinada tRNA. No primeiro passo, a enzima catalisa a ligação covalente de AMP a pelo amino ácido, obtendo-se até ao aminoácido ativado. No segunda passo, o aminoácido ativado é ligado ao tRNA apropriado.
o ATP é hidrolisado, e AMP se anexa ao amino ácido, e pirofosfato é liberado. Durante o segundo passo, o tRNA correto se liga a sintetase. O aminoácido se liga covalentemente à extremidade 3' da tRNA na haste aceitante, e AMP é liberado. Finalmente, o tRNA com seu aminoácido ligado é libertado da enzima. Nesse estágio,
o tRNA é chamado um tRNA carregado ou uma aminoacil-tRNA. Em uma molécula tRNA carregada, o aminoácido é ligado à extremidade 3' do tRNA por uma ligação covalente ( veja a inserção na Figura 1,25).
A capacidade das aminoacil-tRNA-sintetases de reconhecer tRNAs por vezes tem sido chamado de "segundo código genético." Este processo de reconhecimento é necessário para manter a fidelidade da informação genética. A freqüência de erro para aminoacil-tRNA sintetases é inferior a 10^5. Em outras palavras, o amino ácido errado está ligado a um tRNA é menos de uma vez em 100.000 vezes! Como poderia-se esperar, a região do anticódon do tRNA é normalmente importante para o reconhecimento preciso pela aminoacil-tRNA sintetase correta. Em estudos de sinthetases na bactéria Escherichia coli , 17 das 20 tipos de aminoacil-ARNt-sintetases reconhecem a região do anticódon do tRNA. No entanto, outras regiões do tRNA são igualmente reconhecidas. Os locais de reconhecimento são importantes. Estes incluem a haste aceitadora e
bases nas regiões haste de laço.
O tRNA e ATP encaixam precisamente no sítio ativo da enzima, e a estrutura é configurada e concebida para funcionar de uma forma finamente sintonizada. Como poderia um dispositivo tão funcional ser o resultado de forças aleatórias não guiados e reações químicas sem um objetivo final? Como o encaixe preciso para cada uma das vinte amino ácidos poderia ter surgido aleatoriamente, assim como o encaixe para a molécula ATP, que é indispensável para fornecer a energia necessária para a reação poder ocorrer ? Ademais, a aminoacil-tRNA-sintetases é muito específica para esta função, e não tem função nenhuma por si só, mas apenas, se devidamente encaixada no lugar certo para exercer a função especifica com exatidão.
Descasamentos que seguem a Regra Wobble podem ocorrer na terceira posição de emparelhamento no códon-anticódon
Depois de considerar a estrutura e função de moléculas de tRNA, vamos reexaminar algumas características sutis do código genético. O código genético é degenerado, o que significa que mais do que um códon pode especificar o mesmo aminoácido. Degeneração geralmente ocorre na terceira posição do códon. Por exemplo, valina é especificada por GUU, GUC, GUA, e GUG. Em todos os quatro casos, as primeiras duas bases são G e U. A terceira base, no entanto, pode ser U, C, A, ou G. Para explicar esse padrão de degeneração, Francis Crick propôs em 1966 que é devido a "oscilação" na terceira posição no processo de reconhecimento códon-anticódon. Segundo às regras de oscilação, nas duas posições, o primeiro par é estritamente de acordo à regra AU/GC . No entanto, a terceira posição pode tolerar certos tipos de descasamentos (Figura 1.27)

Figura 1.27 Posição Wobble e regras de emparelhamento de bases
(a) A posição oscilante ocorre entre a primeira base (ou seja, a primeira base na direção 5 'para 3') no
anticódon e a terceira base no códon do mRNA.
(b) As bases padrão encontradas no RNA são G, C, A, e U. Além disso, as estruturas das bases de tRNA podem ser modificadas. Algumas bases modificadas que podem ocorrer na posição de oscilação na tRNA são I = inosina; xm5s2U = 5-metil- 2-tiouridina; xm5Um = 5-metil-2'-O-metiluridina; Hum= 2'-O-metiluridina; xm5U = 5-metiluridina; xo5U =5-hydroxyuridine; K2C = lysidine (um derivado de citosina). As bases de mRNA em parênteses são muito mal reconhecidas pelo tRNA.
Por causa das regras de oscilação, é observada uma certa flexibilidade no reconhecimento entre um códon e anticódon durante o processo de tradução. Quando dois ou mais tRNA's que diferem na base oscilante são capazes de reconhecer o mesmo códon, estes são denominados isoacceptor tRNA. Como um exemplo, os tRNA's
com um anticódon de 3'-5'-CCA ou CCG-3'-5' podem reconhecer um codon com a sequência de 5'-3'-GGU. Além disso, a oscilação normas permitem que um único tipo de tRNA possa reconhecer mais que um códon. Por exemplo, um tRNA com uma sequência anticódon de 3'-5'-AAG pode reconhecer um 5'-UUC-3'-5'e um UUU-3'códon. O códon 5'-UUC-3'é uma combinação perfeita com este tRNA. O 5'-UUU-3'códon é incompatível de acordo com a norma. Regras de RNA-RNA de hibridação (ou seja, G no anticódon é incompatível para U no códon), mas os dois podem encaixar de acordo com a as regras de oscilação descritas na Figura 1.27. Da mesma forma, a modificação da base de oscilação para uma inosina permite reconhecer um tRNA de três codons diferentes. Ao nível celular, a capacidade de um tRNA único de reconhecer mais que um códon torna desnecessário de uma célula fazer 61 diferentes moléculas de tRNA com anticódons que são complementares às 61 possíveis códons sentidos. Células de E. coli, por exemplo, produzem uma população de moléculas de tRNA que tem apenas 40 diferentes sequências anticódon.
Moléculas de RNA podem dobrar em estruturas tridimensionais precisas, e as moléculas de tRNA fornecem um exemplo marcante. Quatro segmentos curtos das tRNA dobradas são de dupla hélice, a produção de uma molécula que se parece com uma folha de trevo quando desenhado esquematicamente. Veja abaixo:

Figura 1.25 Estrutura Secundária de tRNA. A numeração convencional dos nucleotídeos inicia na extremidade 5 ' e procede para a extremidade 3 '. Em todos os tRNA's, os nucleotídeos na extremidade 3 'contêm a sequência CCA. Certos locais podem ter nucleótideos adicionais não encontrados em todas as moléculas de tRNA. Estes sítios variáveis são mostrados em azul. A figura também mostra as localizações de poucas bases modificadas especificamente encontradas num tRNA de levedura que transporta alanina. As bases modificadas são de seguinte forma: I = inosina, MI = metilinosina, T = ribothymidine, UH2 =dihydrouridine, M2G = dimethylguanosine, e P = pseudouridina. A inserção mostra um aminoácido ligado de forma covalente à extremidade 3 'de um tRNA.

O reconhecimento do códon é ditado apenas pelo anticódon no tRNA; a estrutura química do aminoácido ligado ao tRNA não desempenha nenhum um papel.

Uma molécula de tRNA. Um tRNA específico para o aminoácido fenilalanina (Phe) é ilustrado de várias maneiras. (A) A estrutura na forma de folha de trevo mostrando a complementaridade do pareamento de bases (linhas vermelhas) que cria as regiões de dupla-hélice na molécula. O anticódon é a sequência de três nucleotídeos que forma pares de bases com o (ódon no mRNA. O aminoácido correspondente ao par códon-anticódon é ligado na extremidade 3' do tRNA. Os tRNAs contêm algumas bases incomuns, as quais são produzidas por modificação química após a síntese do tRNA. Por exemplo, as bases identificadas como 'I' (pseudouridina) e D (diidrouridina) são derivadas de uracila. (B e C) Vistas da molécula em forma de L, com base em análise de difração de raios X. Embora este diagrama ilustre um tRNA para o aminoácido fenilalanina, todos os outros tRNAs têm estruturas semelhantes. (D) A sequência nucleotídica linear da molécula, colorida de acordo com (A), (B) e (C). (E) A representação do tRNA usada neste livro.
Por exemplo, uma sequência 5 "-GCUC-3" em uma parte de uma cadeia de um polinucleótido pode formar uma associação relativamente forte com um "-GAGC-3" sequência 5 na outra região da mesma molécula de tRNA. A folha de trevo é submetida a dobragem adicional, para formar uma estrutura em forma de L compacta que é mantida unida por ligações de hidrogênio adicionais entre as diferentes regiões da molécula. Duas regiões de nucleótidos desemparelhados situadas de um lado da molécula em forma de L são cruciais para a função do tRNA na síntese de proteínas. Uma destas regiões constitui o anticódon, um conjunto de três nucleótidos consecutivos que forma o par ( chave e fechadura ) com o códon complementar de uma molécula de mRNA. A outra é uma região única curta de cadeia simples no "fim da molécula , isto é o local onde o aminoácido que corresponde ao códon se liga ao tRNA. O código genético é redundante;. Isto é, vários códons diferentes podem especificar um único aminoácido. Esta redundância implica que existam mais de um tRNA para muitos dos aminoácidos ou que algumas moléculas de tRNA podem formar um par ( chave na fechadura ) com bases, com mais do que um códon. De fato, ambas as situações ocorrem. Alguns aminoácidos tem mais de um tRNA e alguns tRNA's são construídos de modo que eles necessitam de um emparelhamento de bases preciso apenas com as duas primeiras posições do códon e podem tolerar uma incompatibilidade (ou oscilar) na terceira posição.
A edição por tRNA-sintetases assegura a exatidão
Vários mecanismos trabalhando em conjunto asseguram que a tRNA-sintetase ligue o aminoácido correto a cada tRNA. A sintetase deve inicialmente selecionar o aminoácido correto e o faz, principalmente, por meio de um mecanismo composto de duas etapas. Primeiro, o aminoácido correto apresenta uma maior afinidade pela fenda do sítio ativo de sua sintetase, sendo assim favorecido sobre os outros 19. Em particular, os aminoácidos maiores do que o correto são efetivamente excluídos do sítio ativo. No entanto, a discriminação exata entre dois aminoácidos semelhantes, como a isoleucina e a valina (os quais diferem apenas por um grupo metil), é muito difícil de ser alcançada por um mecanismo de reconhecimento de etapa única. Uma segunda etapa de discriminação ocorre após o aminoácido ter sido ligado covalentemente ao AMP (Figura 1.28)

Figura 1.28 Ativação do aminoácido.
Um aminoácido é ativado para a síntese proteica por uma enzima aminoacil-tRNA-sintetase em duas etapas. Como indicado, a energia da hidrólise de ATP é usada para ligar cada aminoácido a sua molécula de tRNA em uma ligação altamente energética. ° aminoácido é inicialmente ativado por meio da ligação de seu grupo carboxila diretamente a um AMP, formando um aminoácido adenilado; a ligação do AMP, normalmente uma reação desfavorável, é dirigida pela hidrólise da molécula de ATP que doa o AMP. Sem deixar a enzima sintetase, o grupo carboxila ligado ao AMP no aminoácido é, então, transferido para um grupo hidroxila no açúcar, na extremidade 3' da molécula de tRNA. Essa transferência liga o aminoácido por meio de uma ligação éster ativada ao tRNA, formando a molécula
final de aminoacil-tRNA. A enzima sintetase não é mostrada neste diagrama.
Quando o tRNA liga-se à sintetase, ele empurra o aminoácido para uma segunda fenda na sintetase, cujas dimensões precisas excluem o aminoácido correto, mas permitem o acesso de aminoácidos intimamente relacionados. Uma vez que um aminoácido entre nessa fenda de edição, ele será hidrolisado do AMP (ou do próprio tRNA, caso a ligação aminoacil-tRNA já tenha sido formada) e será liberado da enzima. Essa edição hidrolítica, a qual é análoga à correção exonucleotídica mediada pelas DNA-polimerases (Figura 1.29) eleva a exatidão média da taxa de carregamento de tRNA para aproximadamente um erro a cada 40 mil acoplamentos.
Alta fidelidade
Aminoacil-ARNt sintetases devem executar as suas tarefas com alta precisão. Cada erro resultará num aminoácido deslocado quando novas proteínas são construídos. Essas enzimas fazem apenas um erro em 10.000. Para a maioria dos aminoácidos, este nível de precisão não é muito difícil de alcançar. A maioria dos aminoácidos são muito diferentes uns dos outros, e, muitas partes das diferentes tRNA's são utilizadas para reconhecimento preciso. Mas em alguns casos, é difícil escolher apenas os aminoácidos corretos e estas enzimas devem recorrer a técnicas especiais.
Isoleucina é particularmente difícil de reconhecer. É reconhecida por um furo em forma de isoleucina na enzima, o qual é muito pequeno para caber aminoácidos maiores como metionina e fenilalanina, é muito hidrófobo, para ligar qualquer coisa com cadeias laterais polares. Mas, a pouco menor aminoácido valina, diferente por apenas um único grupo metilo, também se encaixa muito bem neste bolso, a ligação em vez de isoleucina ocorre cerca 1 em cada 150 vezes. É demasiado muitos erro, por isso devem ser tomadas medidas corretivas. Isoleucil-tRNA sintetase resolve este problema com um segundo sítio ativo, que realiza uma reação de edição. Isoleucina não se encaixa neste sitio, mas valina errante o faz. O erro é então clivadao, deixando o tRNA pronto para um aminoácido leucina devidamente colocado. Esta etapa de correção melhora a taxa de erro global de cerca de 1 em 3.000.
Esta é uma técnica incrível de revisão de erro, que se junta a outros incríveis mecanismos super elaborados de reparo na célula. Mais uma vez surge a pergunta: Como é que estas máquinas moleculares precisas poderiam ter surgido por meios naturais, sem inteligência envolvido? Isto parece-me mais um incrível exemplo de nano maquinaria molecular altamente sofisticada projetada para desempenhar a sua missão com alto grau de fidelidade e minimização de erros, o que só pode surgir pela previdência de um criador incrivelmente inteligente.

Figura 1.29 Edição hidrolítica. (A) A tRNA-sintetase remove seus próprios erros de pareamento por meio da edição hidrolítica de aminoácidos incorporados incorretamente. O aminoácido correto é rejeitado pelo sitio de edição. (B) ° processo de erro correção realizado pela DNA-polimerase apresenta algumas similaridades; entretanto, difere no que diz respeito ao processo de remoção, o qual depende fortemente de um erro de pareamento com o molde.
Papel crucial das enzimas Aminoacil-tRNA sintetases que ativam o tRNA
Para tentar explicar a origem do código genético, vários pesquisadores têm procurado algum tipo de afinidade química entre os aminoácidos e seus códons correspondentes. Mas esta abordagem é equivocada:
Em primeiro lugar, o código é mediado por tRNA que tem o anticódon (do mRNA) em vez do próprio códon (no DNA). Portanto, se o código fosse baseado em afinidades entre os aminoácidos e os anticódons, isso implicaria que o processo de tradução, através de transcrição não poderia ter surgido em uma segunda fase ou melhoria em um sistema direto mais simples - o complexo processo de duas etapas seria necessário ter surgido desde o início.
Em segundo lugar, o aminoácido não tem um papel na identificação do tRNA ou o códon. Esta associação é feita pela enzima de ativação Aminoacil-ARNt sintetase, que atribui a cada um dos aminoácidos o tRNA apropriado (que requer claramente a enzima identificar corretamente os dois componentes). Existem diferentes enzimas de ativação, 20 - um para cada tipo de aminoácido.
Curiosamente, o lugar do tRNA à qual o aminoácido é anexado tem a mesma sequência nucleotídica para todos os aminoácidos - que constitui um terceiro motivo.
O interesse pelo código genético tende a concentrar-se sobre o papel do tRNA, mas como apenas mencionado, isso é apenas a metade da tarefa de implementação do código para formar proteínas. Tão importante como o emparelhamento códon -anticódon (entre mRNA e tRNA) é a capacidade de cada uma das enzimas de ativação de reunir e anexar cada aminoácido ao seu tRNA apropriado. É evidente que a aplicação do código requer dois conjuntos de moléculas intermediárias: os tRNAs que interagem com os ribossomos e reconhecem o códon apropriado no mRNA, e as enzimas de ativação, Aminoacil-ARNt sintetases, que ligam e anexam os aminoácidos diretamente ao tRNA correto mediante o encaixe preciso e correto ( como se fosse chave-fechadura) . Este é o tipo de complexidade que permeia os sistemas biológicos, e que representa um desafio formidável para uma explicação naturalista de passo a passo mediante eventos apenas baseados em afinidade quimica, ou sorte, para a sua origem. Seria improvável o suficiente se o código foi implementado apenas pelos tRNAs que têm de 70 a 80 nucleotídeos; mas o papel igualmente crucial e complementar os amino ácidos corretos com das enzimas de ativação, que tem centenas de aminoácidos de comprimento. Isto exclui qualquer possibilidade realista de que esse tipo de arranjo poderia ter surgido de forma oportunista, de " boa sorte ", ou acaso. Evolução não seria também um mecanismo viável neste estagio, pois a evolução depende desse processo para começar a ocorrer. Estes mecanismos tinham que surgir antes da primeira célula começar a se auto-replicar.
O desenvolvimento progressivo do código genético não é realista
Tendo em vista os muitos componentes envolvidos na implementação do código genético, pesquisadores da origem da vida tentaram ver como ele poderia ter surgido de forma gradual, de maneira evolutiva. Por exemplo, é geralmente sugerido que o código genético começou com a aplicação de apenas alguns aminoácidos, que, em seguida, gradualmente aumentaram em número. Mas esse tipo de cenário encontra todos os tipos de dificuldades com algo tão fundamental como o código genético.
Em primeiro lugar, afigura-se que os primeiros códons precisavam ter usado apenas duas bases (que pode codificar até 16 aminoácidos); mas uma alteração posterior a três bases (para acomodar 20) iria perturbar gravemente o código. Reconhecendo esta dificuldade, a maioria dos investigadores assumiram que o código utilizava códons de 3 bases, desde o início; que foi notavelmente fortuito ou implicaria em alguma medida de previsão por parte da evolução (o que, evidentemente, não é permitido).
Muito mais grave são as implicações para as proteínas com base em um conjunto severamente limitado de aminoácidos. Em particular, se o código foi limitado a apenas alguns aminoácidos, em seguida, deve-se presumir que as enzimas de ativação composto apenas esse conjunto limitado de aminoácidos, e ainda tinham o nível necessário de especificidade para a implementação segura do código. Não há nenhuma evidência disso; e subsequente a reorganização das enzimas que fariam uso de aminoácidos recentemente disponíveis exigiria mudanças altamente improváveis em sua configuração. Limitações semelhantes se aplicaria aos componentes proteicos dos ribossomos, que têm um papel igualmente essencial na tradução.
Além disso, os tRNA's tendem a ter bases atípicas que são sintetizadas da maneira usual, mas posteriormente modificadas. Estas modificações são realizadas por enzimas, de modo que estas enzimas também teriam de ter começado a vida com base num número limitado de aminoácidos; ou tem de se supor que essas modificações são refinamentos posteriores - mesmo que eles parecem ser necessários para a execução segura do código.
Finalmente, o que vai motivar a adição de novos aminoácidos para o código genético? Eles teriam pouca ou nenhuma utilidade até incorporados em proteínas - mas isso não iria acontecer até que fossem incluídas no código genético. Assim, os novos aminoácidos deve ser sintetizados e de alguma forma incorporados em proteínas úteis (por enzimas que eles não têm), e toda a maquinaria necessária para incluí-los no código (tRNAs dedicados e ativando enzimas) postas em prática - e tudo feito de forma oportunista! Totalmente incrível!
O que deve ser explicado, é o arranjo dos códons na tabela códons padrão que é altamente não aleatório, e serve para traduzir a informação genetica na sequência de aminoácidos para fazer proteínas, bem como a origem da atribuição dos 64 codões tripleto para o 20 aminoácidos. Isto é, a origem da sua tradução. A origem de um alfabeto através dos códons triplet é uma coisa, mas em cima, tem que ser traduzido para um outro "alfabeto", constituído pela sequência de 20 aminoácidos. Isto é, como explicar a origem da capacidade de traduzir a linguagem Inglês para o chinês. Em cima disso, a origem da própria máquina para promover o processo em si tem também que ser explicada, que é o hardware. Quando os seres humanos traduzem Inglês para o chinês, por exemplo, nós reconhecemos a palavra Inglês, e o tradutor conhece o símbolo chinês equivalente e o escreve. Na célula, enzimas Aminoacil-ARNt-sintetase reconhecem o anticódon do tRNA tripleto, e anexam o aminoácido equivalente correto ao tRNA, formando assim cadeias proteicas, que no final faz surgir um polipeptídeo, que se dobra em uma proteína funcional. Como poderiam reações químicas aleatórias ter produzido esse reconhecimento?
Se um codon/anticodon/tRNA não for atribuído ao Aminoacil-ARNt-sintetase correto, o que aconteceria? Vamos supor que o códon AAA em vez de ser atribuído ao amino ácido lisina, for atribuido ao amino ácido aspargina, a proteína vai dobrar de forma errada, ou não vai dobrar, e não vai ser funcional, e vai para a proteassoma, a lixeira celular. O que você acha, como surgiu a atribuição correta do código genético de códons aos amino ácidos corretos ? Se você tiver o alfabeto, e precisar fazer a tradução para o chines, será que com tentativa/erro dos caracteres em chinês, você um dias vai conseguir formular uma frase compreensível em chinês ? as possibilidades são inúmeras, praticamente infinitas. E enquanto a atribuição correta não estava surgindo, a célula ficava esperando num cantinho tomando um Whisky ? Algumas teorias tentam explicar o mecanismo, mas todas elas continuam a ser insatisfatórias. Obviamente. Além disso, Aminoacil-ARNt-sintetase são enzimas complexas.
Por que razão é que elas teriam surgidas, se a função final só poderia ser exercida depois de todo o processo de tradução tiver sido implantado no local, com um ribossomo totalmente funcional ser capaz de fazer o seu trabalho? Lembrando a situação catch22, uma vez que pelo próprio ribossomo é feito o processo próprio em questão? Por que não é racional concluir que o próprio código, o software, bem como o hardware, sua origem são melhor explicados por meio da invenção de um ser extraordinariamente inteligente, em vez de afinidades e reações químicas aleatórias? Perguntas: que utilidade teria o ribossomo sem o tRNA ? sem aminoácidos ? - que são o produto de processos químicos evias metabólicas extremamente complexos ? Que utilidade teria a maquinaria toda, se o código não estivesse estabelecido ( também codificando a construção das maquinas como os ribossomos em questão ? ) , e se nem a atribuição de cada códon ao respectivo aminoácido estivesse presente ? tinha o software e o hardware não ter que estar no lugar ao mesmo tempo?
Haviam todas as partes não ter que estar totalmente funcional plenamente desenvolvidas, entreligados, completamente montados, e ajustados para exercer seu trabalho com precisão como um relógio? E mesmo que vamos supor, as peças estariam totalmente funcionais e no lugar, que vantagem teria isso, se todas as outras peças necessárias não estariam no lugar também, isto é, a dupla hélice do DNA, a sua compactação através de histonas e cromatinas e cromossomos, o seu mecanismo altamente complexo de extração de informação e a transcrição em mRNA? Tinha todo o processo, que é a iniciação da transcrição,o alongamento, o splicing, poliadenilação e terminação, exportação do núcleo para o citosol, a iniciação da síntese de proteínas (TRADUÇÃO), a conclusão da síntese de proteína e DOBRADURA correta das proteínas mediante chaperones , e sua respectiva máquinaria não ter que estar toda no lugar? Será que isso não constitui um complexo sistema interdependente, e irredutível?
Um aspecto raramente discutido de complexidade é como os tRNA's são designados para os aminoácidos corretos. Para o idioma de DNA possa ser traduzido adequadamente, cada anticódon tRNA deve ser ligado ao aminoácido correto. Se este passo crucial na replicação do DNA não for funcional, então a linguagem do DNA não serve para nada. As enzimas aminoacil tRNA sintetases (aaRSs) garantem que o aminoácido apropriado é ligado a um tRNA com o anticódon correto através de uma reação química chamada ' aminoacylação". Tradução precisa exige não só que cada tRNA seja atribuído ao aminoácido correto, mas também que ele não seja aminoacilado por qualquer uma das moléculas de aminoacil tRNA sintetases (aaRSs) dos outros 19 aminoácidos. Um livro de bioquímica observa que, porque todos os aaRSs catalisam reações semelhantes sobre várias moléculas de tRNA semelhantes, pensava-se que "evoluiram de um ancestral comum e devem, portanto, ser estruturalmente relacionados." (Voet e Voet pg. 971-975) No entanto, este não e o caso como "aaRSs formamd um grupo diversificado de [mais de 100] enzimas ... e há pouca semelhança de sequência entre synthetases específicas para os diferentes aminoácidos." (Voet e Voet pg 971-975). Surpreendentemente, estes próprios aaRSs são codificados pelo DNA: isto constitui a essência de um problema de ovo de galinha. As enzimas a construir ajudam a realizar a mesma tarefa que as constrói!
tradução de:
http://reasonandscience.heavenforum.org/t2057-origin-of-translation-of-the-4-nucleic-acid-bases-and-the-20-amino-acids-and-the-universal-assignment-of-codons-to-amino-acids#3528

